
AlcheSMILES
- Tôi dùng IDA để đọc code của bài này.
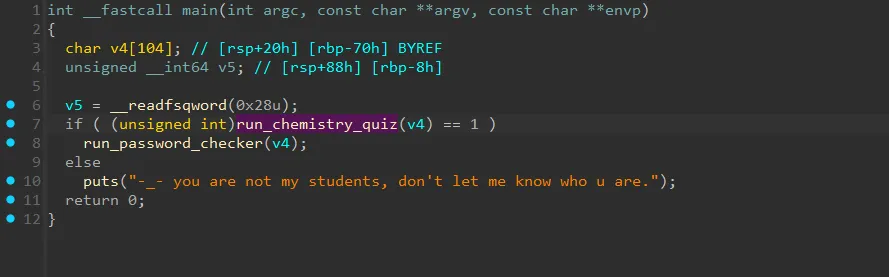
- Ở hàm main này thì khá tường minh và có thể đoán ngay chương trình sẽ đi qua 1 hàm các câu hỏi là
run_chemistry_quizvà một hàm check password làrun_password_checker. - Trước tiên hãy cùng phân tích xem hàm
run_chemistry_quizđã làm gì.
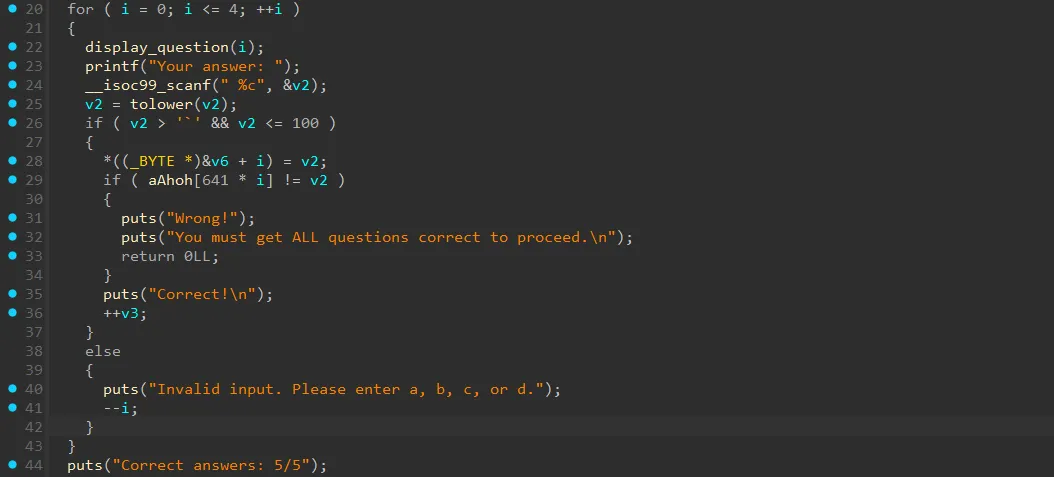
- Ở vòng for đầu tiên thì đang thực hiện check các đáp án (a/b/c/d) mà người dùng nhập vào cho các câu hỏi hóa học.
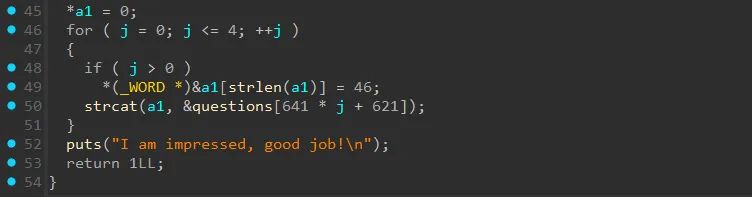
- Tiếp đến, ở vòng for thứ 2 chương trình sẽ gán các đáp án đúng vào biến
a1, mỗi đáp án cách nhau bởi dấu.. - Đây là kết quả của
a1sau khi kết thúc hàmrun_chemistry_quiz.
HOH.HOH.CCO.c1ccc(O)cc1.CCNC- Bây giờ ta sẽ phân tích tiếp hàm
run_password_checker.
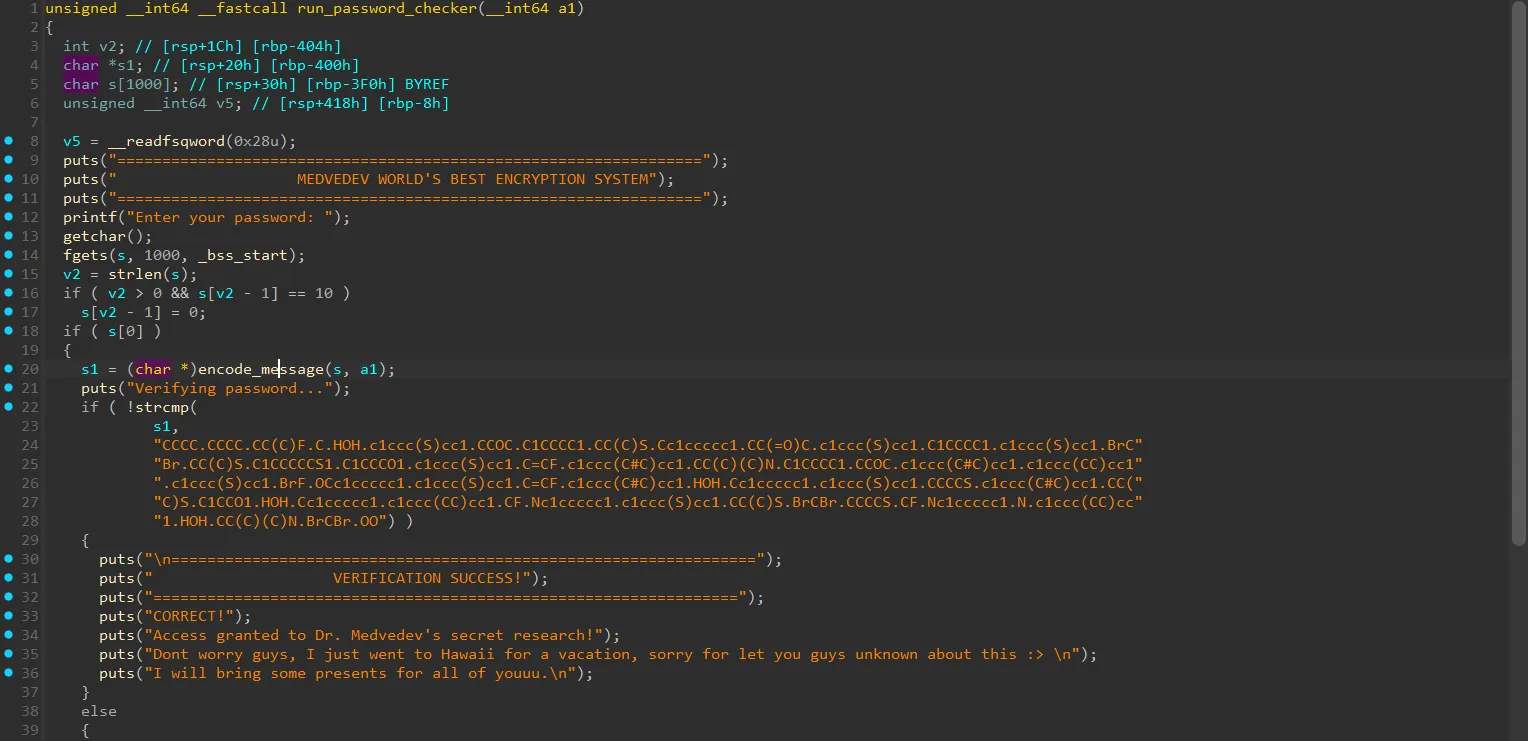
- Hàm này chỉ đơn giản là cho người dùng nhập vào 1 chuỗi, sau đó đi qua hàm
encode_messageđể mã hóa và cuối cùng là so sánh chuỗi đã được mã hóa với dữ liệu có sẵn của chương trình. - Ta cùng check xem hàm
encode_messageđang làm gì. - Trước hết hàm này sẽ đi qua hàm
create_permutationđể hash key và gán vào mảngv9, dưới đây là phiên bản python tường minh hơn của hàmcreate_permutation.
def hash_molecular_key(s): v2 = 0 for c in s: v2 = 31 * v2 + ord(c) return v2 & 0x7FFFFFFF
def create_permutation(key, v9): v4 = hash_molecular_key(key) for j in range(127, 0, -1): v4 = (16807 * v4) % 0x7FFFFFFF idx = v4 % (j + 1) v9[j], v9[idx] = v9[idx], v9[j]- Tiếp đến đi qua vòng for mapping ngược các ký tự ở nửa sau của v9.
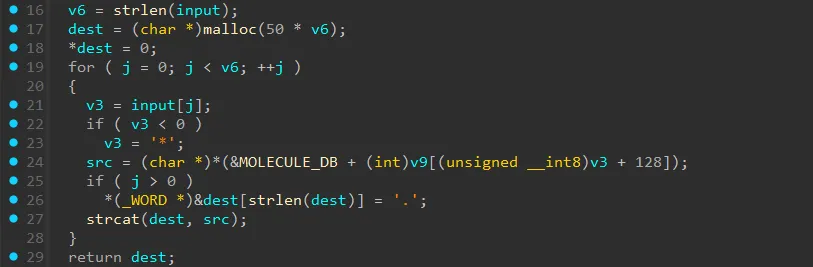
- Đây chính là hàm xử lý chính của input nhập vào.
- Vòng for này duyệt qua toàn bộ các ký tự của input.
- Với mỗi ký tự,
srcsẽ được gán với chuỗi trong mảngMOLECULE_DBtương ứng giá trị của v9 với index được tính từ ascii của ký tự đó cộng với 128. - Các chuỗi được cách nhau bởi một dấu
.. - Bây giờ ta đã xác định được toàn bộ mạch logic của chương trình.
- Để dịch ngược lại flag ta cần lấy từng chuỗi trong mảng encoded.
- Sau đó tính index và trừ đi 128 thì ta sẽ có được từng ký tự của flag.
- Dưới đây là script python của tôi để tính và in ra flag cho bài này.
MOLECULE_DB = [ "C", "CC", "CCC", "O", "CO", "CCO", "N", "CN", "CCN", "S", "CS", "CCS", "F", "CF", "CCF", "Cl", "CCl", "CCCl", "Br", "CBr", "HOH", "OO", "OCO", "c1ccccc1", "Cc1ccccc1", "OCc1ccccc1", "Nc1ccccc1", "Sc1ccccc1", "c1ccc(C)cc1", "c1ccc(O)cc1", "c1ccc(N)cc1", "c1ccc(S)cc1", "c1ccc(F)cc1", "CC(C)C", "CC(C)O", "CC(C)N", "CC(C)S", "CC(C)F", "CCCC", "CCCCO", "CCCCN", "CCCCS", "CCCCF", "C1CC1", "C1CCO1", "C1CCN1", "C1CCS1", "C1CCF1", "C=C", "C=CO", "C=CN", "C=CS", "C=CF", "C#C", "C#CO", "C#CN", "C#CS", "C#CF", "c1ccncc1", "c1ccnc(C)c1", "c1ccnc(O)c1", "c1ccnc(N)c1", "c1ccnc(S)c1", "CC=C", "CCC=C", "CCCC=C", "CCCCC=C", "CCCCCC=C", "c1cccnc1", "c1ccc(C=C)cc1", "c1ccc(C#C)cc1", "c1ccc(CC)cc1", "c1ccc(CCC)cc1", "COC", "CCOC", "CCCOC", "CCCCOC", "CCCCCOC", "CNC", "CCNC", "CCCNC", "CCCCNC", "CCCCCNC", "CSC", "CCSC", "CCCSC", "CCCCSC", "CCCCCSC", "CFC", "CCFC", "CCCFC", "CCCCFC", "CCCCCFC", "ClCCl", "BrCBr", "ICl", "ClF", "BrF", "c1ccc2ccccc2c1", "c1ccc2cc(C)ccc2c1", "c1ccc2cc(O)ccc2c1", "c1ccc2cc(N)ccc2c1", "c1ccc2cc(S)ccc2c1", "CC(C)(C)C", "CC(C)(C)O", "CC(C)(C)N", "CC(C)(C)S", "CC(C)(C)F", "C1CCC1", "C1CCCO1", "C1CCCN1", "C1CCCS1", "C1CCCF1", "C1CCCC1", "C1CCCCO1", "C1CCCCN1", "C1CCCCS1", "C1CCCCF1", "C1CCCCC1", "C1CCCCCO1", "C1CCCCCN1", "C1CCCCCS1", "C1CCCCCF1", "c1ccc(C(C)C)cc1", "c1ccc(C(C)O)cc1", "c1ccc(C(C)N)cc1", "CC(=O)C", "CC(=O)O"]
def hash_molecular_key(s): v2 = 0 for c in s: v2 = 31 * v2 + ord(c) return v2 & 0x7FFFFFFF
def create_permutation(key, v9): v4 = hash_molecular_key(key) for j in range(127, 0, -1): v4 = (16807 * v4) % 0x7FFFFFFF idx = v4 % (j + 1) v9[j], v9[idx] = v9[idx], v9[j]
key = "HOH.HOH.CCO.c1ccc(O)cc1.CCNC"encoded = "CCCC.CCCC.CC(C)F.C.HOH.c1ccc(S)cc1.CCOC.C1CCCC1.CC(C)S.Cc1ccccc1.CC(=O)C.c1ccc(S)cc1.C1CCCC1.c1ccc(S)cc1.BrCBr.CC(C)S.C1CCCCCS1.C1CCCO1.c1ccc(S)cc1.C=CF.c1ccc(C#C)cc1.CC(C)(C)N.C1CCCC1.CCOC.c1ccc(C#C)cc1.c1ccc(CC)cc1.c1ccc(S)cc1.BrF.OCc1ccccc1.c1ccc(S)cc1.C=CF.c1ccc(C#C)cc1.HOH.Cc1ccccc1.c1ccc(S)cc1.CCCCS.c1ccc(C#C)cc1.CC(C)S.C1CCO1.HOH.Cc1ccccc1.c1ccc(CC)cc1.CF.Nc1ccccc1.c1ccc(S)cc1.CC(C)S.BrCBr.CCCCS.CF.Nc1ccccc1.N.c1ccc(CC)cc1.HOH.CC(C)(C)N.BrCBr.OO"
v9 = list(range(128)) + [0] * 128create_permutation(key, v9)for i in range(128): v9[v9[i] + 128] = i
flag = ""for m in encoded.split('.'): idx = MOLECULE_DB.index(m) flag += chr(v9[128:].index(idx))print(flag)- Sau khi run code thì đã có được flag cho bài này.
Flag
DDC{1_gu3s5_u_n3v4_7hought_0f_7h1s_ch3m1stry_3ncrypt1on}